# `clickSelects` et `showSelected` nommés
<!-- paragraph -->
```{r setup, echo=FALSE}
knitr::opts_chunk$set(fig.path="ch14-figures/")
```
<!-- paragraph -->
Dans ce chapitre nous expliquons comment utiliser les [variables `clickSelects` et `showSelected` nommées](/ch06#data-driven-selectors) pour créer des noms de sélecteurs basés sur des données.
<!-- comment -->
Cette fonctionnalité facilite l’écriture du code `animint2` et accélère la compilation.
<!-- paragraph -->
Plan du chapitre :
<!-- paragraph -->
- Nous commençons par charger le jeu de données `PSJ` et compiler les données à tracer.
<!-- comment -->
- Nous présentons une méthode pour définir un `animint` avec de nombreux sélecteurs, en utilisant des boucles `for`.
<!-- comment -->
Cette approche est techniquement correcte, mais peu efficace.
<!-- comment -->
- Nous expliquons ensuite la méthode à privilégier pour définir un `animint` avec de nombreux sélecteurs, c’est-à-dire en utilisant des `clickSelects` et `showSelected` nommés.
<!-- comment -->
Cette méthode est plus efficace sur le plan computationnel et plus facile à coder.
<!-- paragraph -->
## Téléchargement du jeu de données {#download}
<!-- paragraph -->
Les données de l'exemple proviennent du [package PeakSegJoint](https://github.com/tdhock/PeakSegJoint) qui détecte les pics dans des séquences de données génomiques.
<!-- comment -->
Le code ci-dessous télécharge le jeu de données.
<!-- paragraph -->
```{r}
if(!requireNamespace("animint2data"))
remotes::install_github("animint/animint2data")
data(PSJ, package="animint2data")
sapply(PSJ, class)
```
<!-- paragraph -->
On peut voir, ci-dessus, que `PSJ` est une liste de plusieurs éléments (listes et tableaux de données).
<!-- paragraph -->
## Exploration des données `PSJ` à l’aide de ggplots statiques {#explore-data-with-static-ggplots}
<!-- paragraph -->
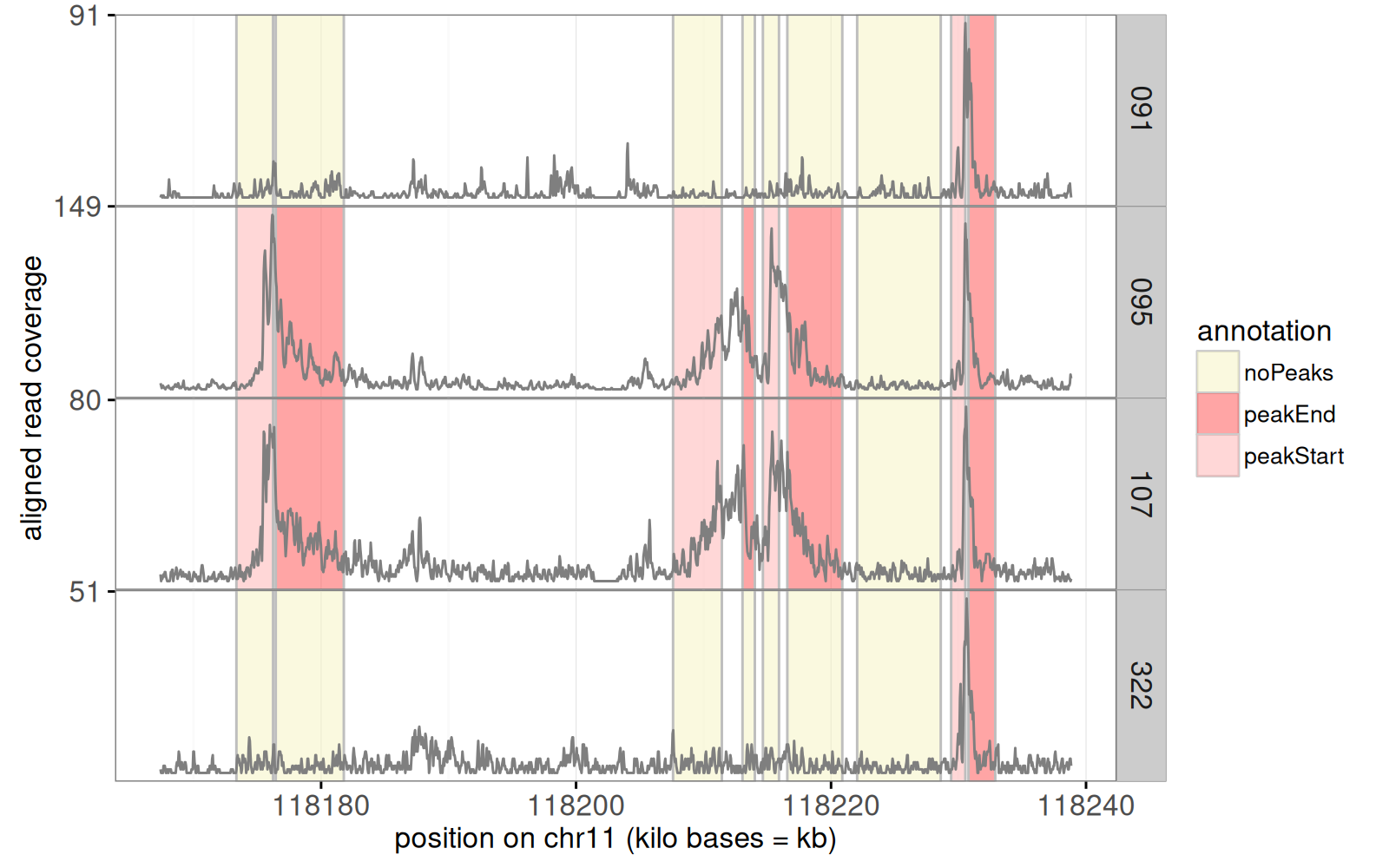
Nous commençons par un graphique de quelques données ChIP-seq génomiques, des données séquentielles dont les valeurs augmentent dans les zones actives (généralement lorsque des gènes sont transcrits).
<!-- comment -->
Dans le code ci-dessous, nous affichons chaque échantillon dans un panneau séparé.
<!-- paragraph -->
```{r}
library(animint2)
ann.colors <- c(
noPeaks="#f6f4bf",
peakStart="#ffafaf",
peakEnd="#ff4c4c",
peaks="#a445ee")
(gg.cov <- ggplot()+
scale_y_continuous(
"couverture des lectures alignées",
breaks=function(limits){
floor(limits[2])
})+
scale_x_continuous(
"position sur chr11 (kilobases = kb)")+
coord_cartesian(xlim=c(118167.406, 118238.833))+
geom_tallrect(aes(
xmin=chromStart/1e3, xmax=chromEnd/1e3,
fill=annotation),
alpha=0.5,
color="grey",
data=PSJ$filled.regions)+
scale_fill_manual(values=ann.colors)+
theme_bw()+
theme(panel.margin=grid::unit(0, "cm"))+
facet_grid(sample.id ~ ., labeller=function(df){
df$sample.id <- sub("McGill0", "", sub(" ", "\n", df$sample.id))
df
}, scales="free")+
geom_line(aes(
base/1e3, count),
data=PSJ$coverage,
color="grey50"))
```
<!-- paragraph -->
La figure ci-dessus montre les données brutes bruitées sous forme d’un `geom_line()` gris.
<!-- comment -->
Les rectangles de couleur indiquent si le début ou la fin d’un pic doit être prédit dans une région et un échantillon donnés.
<!-- comment -->
Ensuite, nous ajoutons un panneau pour les problèmes de segmentation.
Dans chaque problème, un algorithme a cherché un pic commun à tous les échantillons.
<!-- comment -->
Pour chaque problème, l’algorithme prédit les positions de début et de fin du pic de valeur élevée.
<!-- comment -->
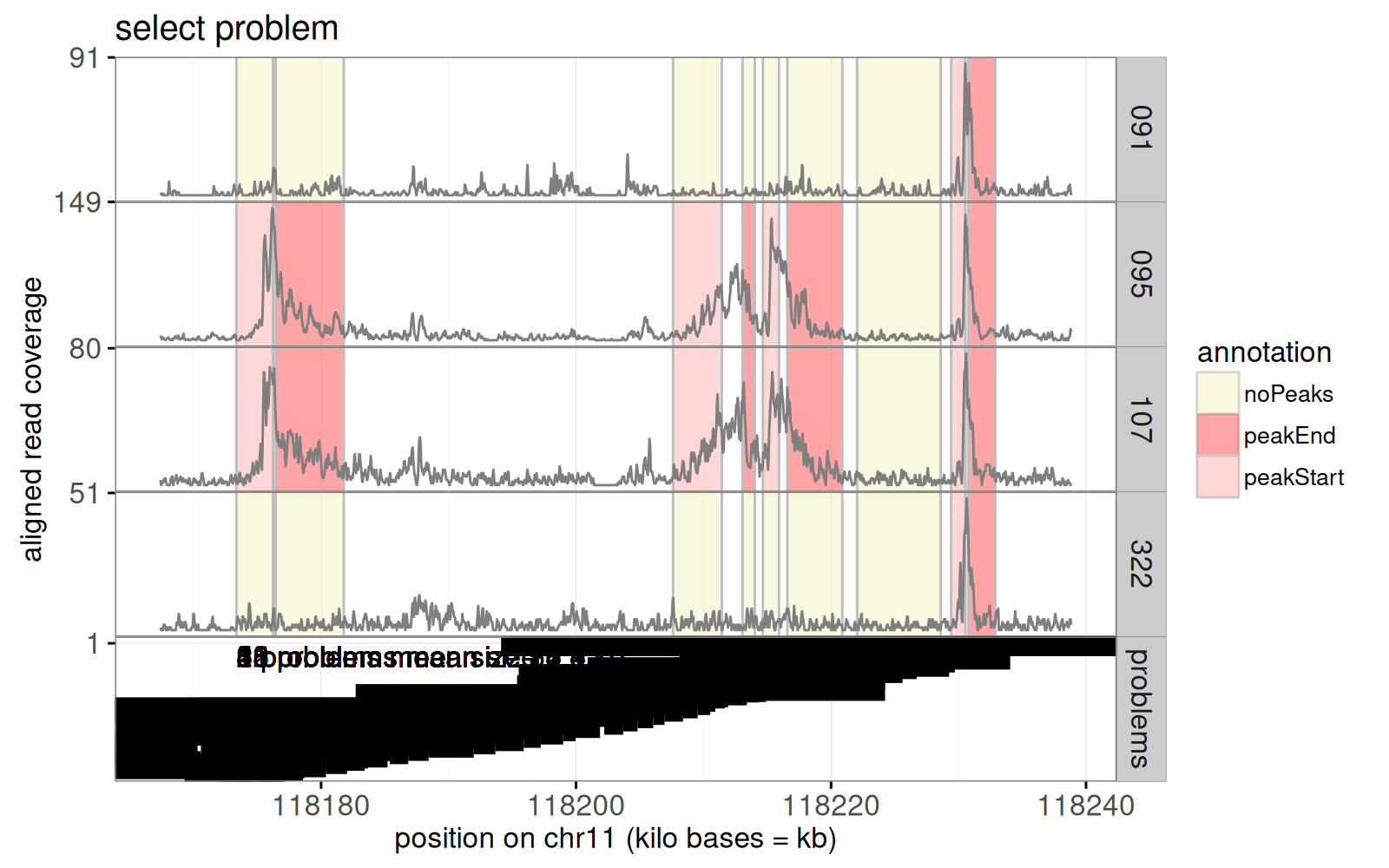
Le code ci-dessous calcule un tableau de contingence avec une ligne pour chaque problème de ce type.
<!-- paragraph -->
```{r}
library(data.table)
(show.problems <- data.table(PSJ$problems)[
, y := problem.i/max(problem.i), by=bases.per.problem][])
```
<!-- paragraph -->
Dans le code ci-dessus une colonne `y` est ajoutée pour l’affichage des problèmes.
<!-- paragraph -->
```{r}
(gg.cov.prob <- gg.cov+
ggtitle("Sélection du problème")+
geom_text(aes(
chromStart/1e3, 0.9,
label=sprintf(
"%d taille moyenne des problèmes %.1f kb",
problems, mean.bases/1e3)),
showSelected="bases.per.problem",
data=PSJ$problem.labels,
hjust=0)+
geom_segment(aes(
problemStart/1e3, y,
xend=problemEnd/1e3, yend=y),
showSelected="bases.per.problem",
clickSelects="problem.name",
size=5,
data=show.problems))
```
<!-- paragraph -->
Ci-dessus, on voit qu’un panneau inférieur intitulé `problems` a été ajouté au graphique précédent.
<!-- comment -->
Le graphique statique ci-dessus est surchargé ; la version interactive sera lisible parce qu’elle n’affichera qu’une seule valeur de `bases.per.problem` à la fois.
<!-- comment -->
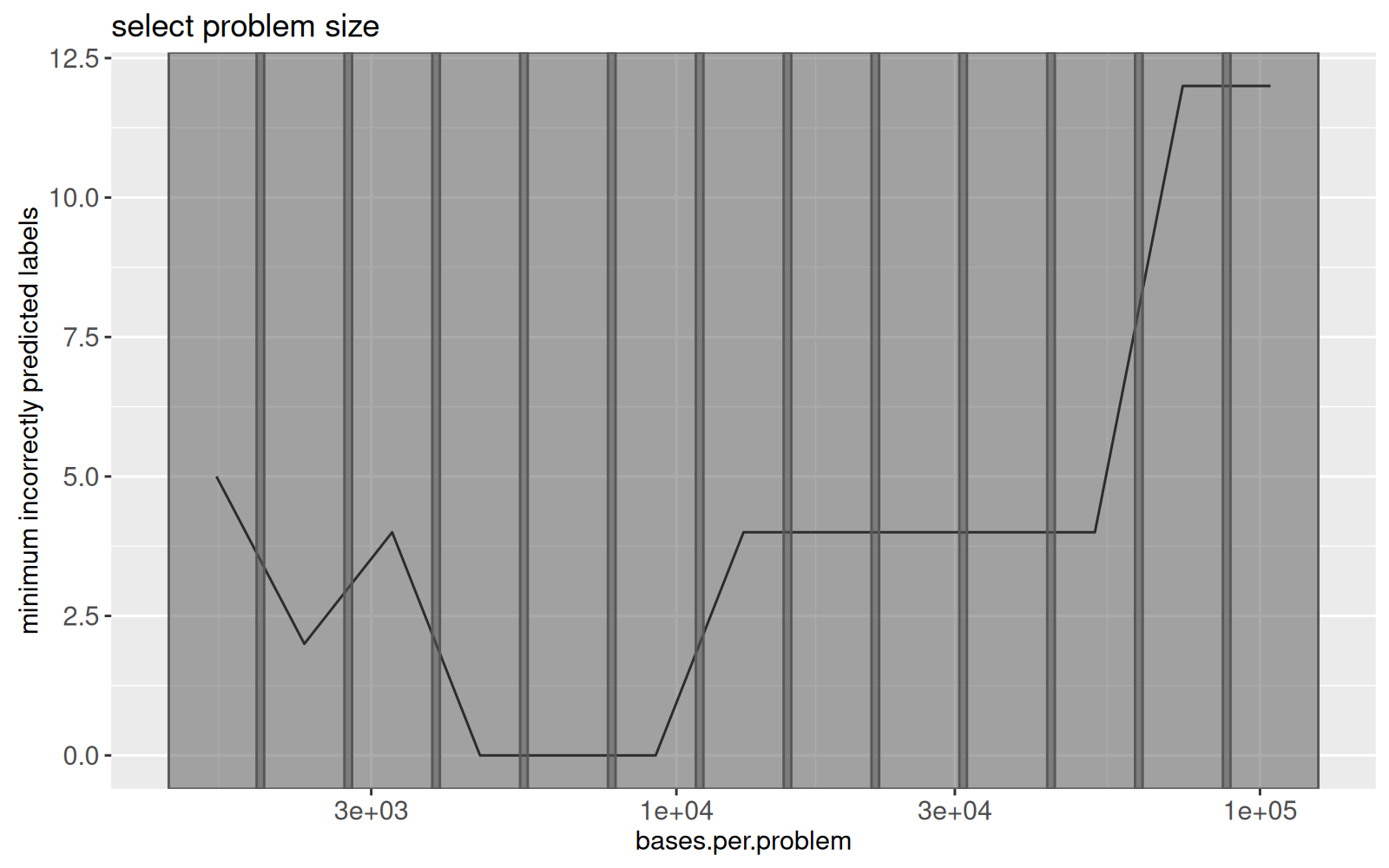
Pour sélectionner les différentes valeurs de `bases.per.problem` (taille du problème), nous utiliserons un autre graphique, qui présentera le meilleur taux d’erreur pour chaque taille de problème, comme le montrent les données ci-dessous.
<!-- paragraph -->
```{r}
(res.error <- data.table(PSJ$error.total.chunk))
```
<!-- paragraph -->
Le tableau de contingence ci-dessus comporte une ligne par valeur de `bases.per.problem`, un paramètre de taille de fenêtre coulissante, que nous allons explorer à l’aide de l’interactivité.
<!-- comment -->
Nous utilisons ces données pour tracer le graphique ci-dessous.
<!-- paragraph -->
```{r}
(gg.res.error <- ggplot()+
ggtitle("choisir la taille du problème")+
ylab("nombre minimal d’erreurs de prédiction d’étiquettes")+
geom_line(aes(
bases.per.problem, errors),
data=res.error)+
geom_tallrect(aes(
xmin=min.bases.per.problem,
xmax=max.bases.per.problem),
clickSelects="bases.per.problem",
alpha=0.5,
data=res.error)+
scale_x_log10())
```
<!-- paragraph -->
La figure ci-dessus montre le nombre minimum d’erreurs d’étiquettes en fonction de la taille du problème.
<!-- comment -->
Des rectangles gris seront utilisés pour sélectionner la taille du problème.
<!-- paragraph -->
Un paramètre de pénalité contrôle le nombre d’échantillons avec un pic commun, il est défini dans le tableau de données de sélection de modèle dans le code ci-dessous.
<!-- paragraph -->
```{r}
pdot <- function(L){
out_list <- list()
for(i in seq_along(L)){
out_list[[i]] <- data.table(
problem.dot=names(L)[[i]], L[[i]])
}
rbindlist(out_list)
}
(all.modelSelection <- pdot(PSJ$modelSelection.by.problem))
```
<!-- paragraph -->
Le code ci-dessus utilise la fonction `pdot()` qui fait appel à la méthode [liste de tableaux de données](/ch99#list-of-data-tables) pour ajouter une colonne nommée `problem.dot`, laquelle servira ensuite à définir les sélecteurs dans la visualisation interactive.
<!-- comment -->
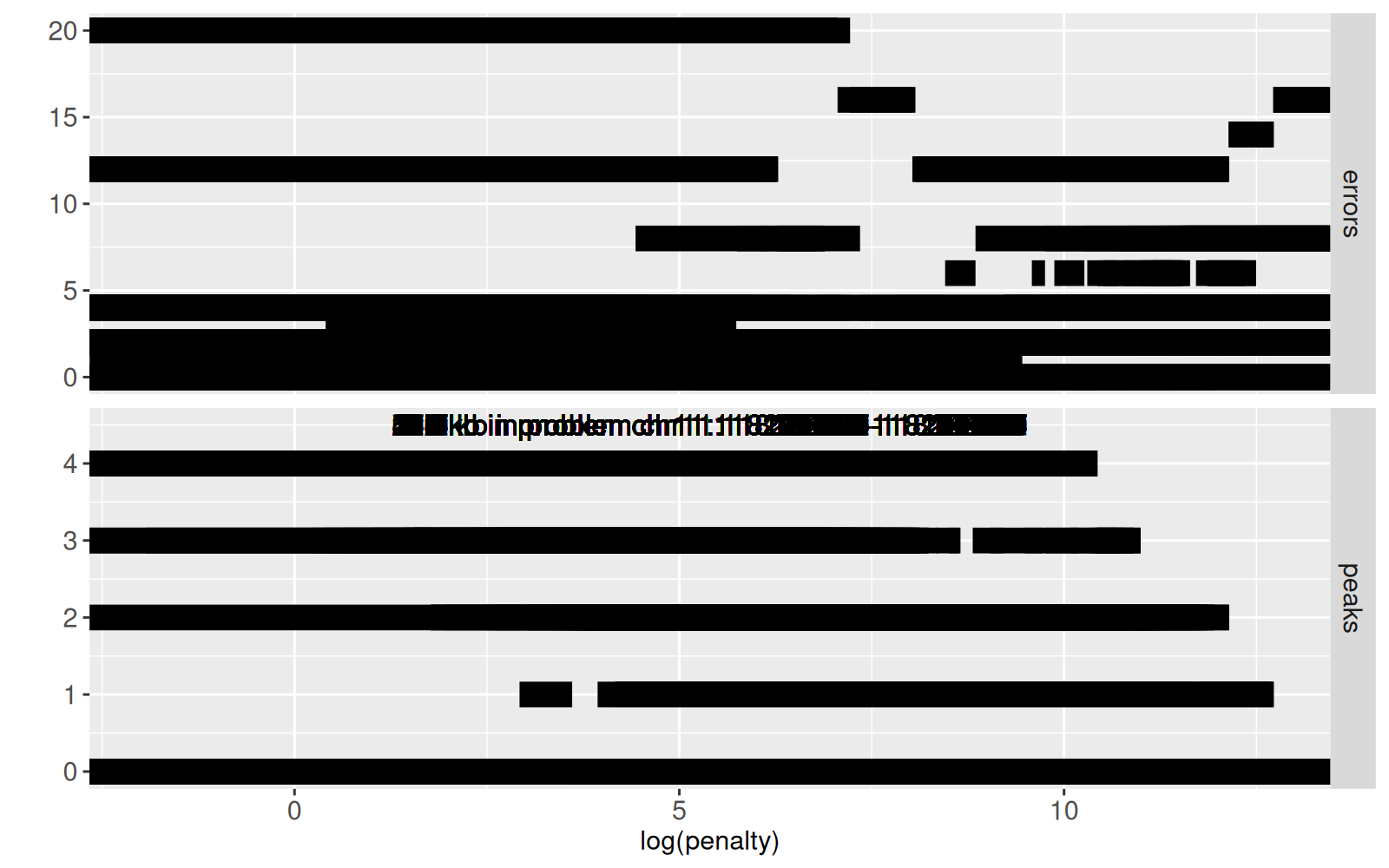
Nous traçons ci-dessous le nombre de pics et d’erreurs d’étiquettes, en fonction du paramètre de pénalité de l’algorithme.
<!-- paragraph -->
```{r}
long.modelSelection <- melt(
data.table(all.modelSelection)[, errors := as.numeric(errors)],
measure.vars=c("peaks","errors"))
log.lambda.range <- all.modelSelection[, c(
min(max.log.lambda), max(min.log.lambda))]
modelSelection.labels <- unique(all.modelSelection[, data.table(
problem.name,
bases.per.problem,
problemStart,
problemEnd,
log.lambda=mean(log.lambda.range),
peaks=max(peaks)+0.5)])
(gg.model.selection <- ggplot()+
scale_x_continuous("log(pénalité)")+
geom_segment(aes(
min.log.lambda, value,
xend=max.log.lambda, yend=value),
showSelected=c("bases.per.problem", "problem.name"),
data=long.modelSelection,
size=5)+
geom_text(aes(
log.lambda, peaks,
label=sprintf(
"%.1f kb dans le problème %s",
(problemEnd-problemStart)/1e3, problem.name)),
showSelected=c("bases.per.problem", "problem.name"),
data=data.frame(modelSelection.labels, variable="peaks"))+
ylab("")+
facet_grid(variable ~ ., scales="free"))
```
<!-- paragraph -->
La figure ci-dessus montre `errors` (panneau supérieur) et `peaks` (panneau inférieur) en fonction de `log(penalty)`.
<!-- comment -->
Là encore, cette version statique est surchargée ; l’interactivité sera utilisée pour rendre la figure lisible (en n’affichant que le sous-ensemble de données correspondant aux valeurs sélectionnées de `bases.per.problem` et `problem.name`).
<!-- paragraph -->
## Visualisation interactive des données (incomplète) {#incomplete}
<!-- paragraph -->
Dans cette section, nous combinons les `ggplots` de la section précédente pour créer une visualisation avec interactivité qui rélie les graphiques.
<!-- comment -->
Le code ci-dessous utilise `theme_animint()` pour associer certaines options d’affichage au graphique de couverture précédent, et l’option `first` est ajoutée pour spécifier quels sous-ensembles de données doivent être affichés en premier.
<!-- paragraph -->
```{r}
timing.incomplete.construct <- system.time({
coverage.counts <- table(PSJ$coverage$sample.id)
facet.rows <- length(coverage.counts)+1
vis.incomplete <- animint(
out.dir="ch14incomplete",
first=list(
bases.per.problem=6516,
problem.name="chr11:118174946-118177139"),
coverage=gg.cov.prob+theme_animint(
last_in_row=TRUE, colspan=2,
width=800, height=facet.rows*100),
resError=gg.res.error,
modelSelection=gg.model.selection)
})
```
<!-- paragraph -->
La sortie temporelle ci-dessus montre que la définition initiale est rapide.
<!-- comment -->
Générer cette visualisation des données préliminaires (incomplètes) dans le code ci-dessous est également rapide.
<!-- paragraph -->
```{r ch14incomplete}
before.incomplete <- Sys.time()
vis.incomplete
cat(elapsed.incomplete <- Sys.time()-before.incomplete, "secondes\n")
```
<!-- paragraph -->
Nous voyons ci-dessus une visualisation des données avec trois graphiques.
<!-- paragraph -->
- Dans la partie supérieure, quatre profils de données ChIP-seq sont affichés, ainsi qu’un panneau de problèmes qui divise l’axe X en sections dans lesquels l’algorithme de segmentation s’exécute.
<!-- comment -->
- En cliquant sur le graphique en bas à gauche, on sélectionne la taille du problème, ce qui met à jour les problèmes affichés en haut.
<!-- comment -->
- Le graphique en bas à droite montre le nombre de pics et d’erreurs en fonction de la pénalité (plus grande pour moins de pics).
<!-- paragraph -->
## Ajouter de l’interactivité à l’aide de boucles `for` {#define-using-for-loops}
<!-- paragraph -->
Dans cette section, nous ajoutons des couches aux `ggplots` précédents en utilisant une boucle `for`, une approche sous-optimale, que nous présentons pour la comparer à une meilleure approche (`clickSelects` et `showSelected` nommés), présentée dans la prochaine section.
<!-- comment -->
La visualisation de la section précédente est incomplète.
<!-- comment -->
Nous aimerions ajouter
<!-- paragraph -->
- des rectangles dans le graphique en bas à gauche pour nous permettre de sélectionner le nombre de pics prédits dans un problème donné ;
<!-- comment -->
- des segments et des rectangles dans le graphique du haut pour montrer les pics prédits et les erreurs d’étiquettes.
<!-- paragraph -->
Une façon (inefficace) d’ajouter ces éléments serait d’utiliser la boucle `for` codée ci-dessous.
<!-- comment -->
Pour chaque problème, il existe un sélecteur (appelé `problem.dot`) pour le nombre de pics dans ce problème.
<!-- comment -->
Dans cette boucle `for`, nous ajoutons donc quelques couches aux graphiques `coverage` et `modelSelection` à l’aide de `clickSelects="problem.dot"` ou `showSelected="problem.dot"` .
<!-- paragraph -->
```{r}
vis.first <- vis.incomplete
vis.first$first <- c(vis.incomplete$first, PSJ$first)
vis.first$modelSelection <- vis.first$modelSelection+
ggtitle("choisir le nombre d’échantillons ayant un pic commun")
print(timing.for.construct <- system.time({
vis.for <- vis.first
vis.for$title <- "PSJ avec boucles for"
vis.for$out.dir <- "ch14for"
for(problem.dot in names(PSJ$modelSelection.by.problem)){
if(problem.dot %in% names(PSJ$peaks.by.problem)){
peaks <- PSJ$peaks.by.problem[[problem.dot]]
peaks[[problem.dot]] <- peaks$peaks
prob.peaks.names <- c(
"bases.per.problem", "problem.i", "problem.name",
"chromStart", "chromEnd", problem.dot)
prob.peaks <- unique(data.frame(peaks)[, prob.peaks.names])
prob.peaks$sample.id <- "problems"
vis.for$coverage <- vis.for$coverage +
geom_segment(aes(
chromStart/1e3, 0,
xend=chromEnd/1e3, yend=0),
clickSelects="problem.name",
showSelected=c(problem.dot, "bases.per.problem"),
data=peaks, size=7, color="deepskyblue")
}
modelSelection.dt <- PSJ$modelSelection.by.problem[[problem.dot]]
modelSelection.dt[[problem.dot]] <- modelSelection.dt$peaks
vis.for$modelSelection <- vis.for$modelSelection+
geom_tallrect(aes(
xmin=min.log.lambda,
xmax=max.log.lambda),
clickSelects=problem.dot,
showSelected=c("problem.name", "bases.per.problem"),
data=modelSelection.dt, alpha=0.5)
}
}))
```
<!-- paragraph -->
Notez le temps d’exécution du code ci-dessus, qui comprend uniquement l’évaluation du code R pour les ggplots (et non l’affichage).
<!-- comment -->
Ensuite, nous compilons la visualisation des données.
<!-- paragraph -->
```{r ch14for}
before.for <- Sys.time()
vis.for
cat(elapsed.for <- Sys.time()-before.for, "seconds\n")
```
<!-- paragraph -->
Notez que la compilation nécessite plusieurs secondes, car il y a de nombreux geoms (cliquez sur « Show download status table » pour les voir tous).
<!-- comment -->
Par rapport à la visualisation des données de la section précédente, celle-ci présente
<!-- paragraph -->
- des segments bleus qui apparaissent dans le graphique des données de couverture supérieure, pour indiquer les pics prédits ;
<!-- comment -->
- des rectangles de sélection qui peuvent être cliqués dans le graphique en bas à droite, pour modifier le nombre d’échantillons présentant un pic dans le problème sélectionné.
<!-- paragraph -->
Dans la prochaine section, nous allons créer la même visualisation des données, mais de manière plus efficace.
<!-- paragraph -->
## Définition de l’interactivité à l’aide de `clickSelects` et `showSelected` nommés {#define-using-named}
<!-- paragraph -->
Dans cette section, nous créons une version plus efficace de la précédente visualisation des données en utilisant des `clickSelects` et des `showSelected` nommés.
<!-- comment -->
En général, toute visualisation des données définie à l’aide de boucles `for` dans le code R peut être optimisée par cette méthode.
<!-- comment -->
Tout d’abord, nous définissons quelques données communes.
<!-- paragraph -->
```{r}
(sample.peaks <- pdot(PSJ$peaks.by.problem))
```
<!-- paragraph -->
La sortie ci-dessus montre un tableau avec une ligne par pic qui peut être affiché, pour différents échantillons, problèmes, et choix interactifs des variables `bases.per.problem` et `peaks`.
<!-- comment -->
Notez la colonne `problem.dot` : elle détermine le nom du sélecteur qui stockera le nombre de pics sélectionné pour le problème.
<!-- paragraph -->
L’idée principale du code ci-dessous est celle-ci : pour chaque problème, un sélecteur est défini par la colonne `problem.dot` pour le nombre de pics dans ce problème.
<!-- comment -->
Nous utilisons `showSelected=c(problem.dot="peaks")` et `clickSelects=c(problem.dot="peaks")` pour indiquer que le nom du sélecteur se trouve dans la colonne `problem.dot`, et que la valeur de la sélection se trouve dans la colonne `peaks`.
<!-- comment -->
Le compilateur `animint2dir()` crée une variable de sélection pour chaque valeur unique de `problem.dot` (et il utilise les valeurs correspondantes dans `peaks` pour définir/mettre à jour la/les valeur/geoms sélectionnée(s)).
<!-- paragraph -->
```{r}
print(timing.named.construct <- system.time({
vis.named <- vis.first
vis.named$title <- "PSJ avec clickSelects et showSelected nommés"
vis.named$out.dir <- "ch14named"
vis.named$coverage <- vis.named$coverage+
geom_segment(aes(
chromStart/1e3, 0,
xend=chromEnd/1e3, yend=0),
clickSelects="problem.name",
showSelected=c(problem.dot="peaks", "bases.per.problem"),
data=sample.peaks, size=7, color="deepskyblue")
vis.named$modelSelection <- vis.named$modelSelection+
geom_tallrect(aes(
xmin=min.log.lambda,
xmax=max.log.lambda),
clickSelects=c(problem.dot="peaks"),
showSelected=c("problem.name", "bases.per.problem"),
data=all.modelSelection, alpha=0.5)
}))
```
<!-- paragraph -->
De toute évidence, le code R ci-dessus, qui utilise les `clickSelects` et `showSelected` nommés, est beaucoup plus rapide.
<!-- comment -->
Nous le compilons ci-dessous.
<!-- paragraph -->
```{r ch14named}
before.named <- Sys.time()
vis.named
cat(elapsed.named <- Sys.time()-before.named, "seconds\n")
```
<!-- paragraph -->
L’`animint` produit ci-dessus devrait être identique à l’autre visualisation des données de la section précédente.
<!-- comment -->
Les temps d’exécution ci-dessus montrent que les `clickSelects` et `showSelected` nommés sont beaucoup plus rapides que les boucles `for`, tant au niveau de la définition que de la compilation.
<!-- paragraph -->
## Comparaison de l’utilisation du disque {#disk-usage}
<!-- paragraph -->
Dans cette section, nous calculons l’utilisation du disque pour les deux méthodes.
<!-- paragraph -->
```{r}
vis.dirs.vec <- c("ch14incomplete", "ch14for", "ch14named")
vis.dirs.text <- paste(vis.dirs.vec, collapse=" ")
(cmd <- paste("du -ks", vis.dirs.text))
(kb.dt <- fread(cmd=cmd, col.names=c("kilobytes", "path")))
```
<!-- paragraph -->
Le tableau de contingence ci-dessus montre que la visualisation des données définie à l’aide de boucles `for` prend environ deux fois plus d’espace disque que la visualisation des données utilisant des `clickSelects` et `showSelected` nommés.
<!-- paragraph -->
## Résumé du chapitre et exercices {#ch14-exercises}
<!-- paragraph -->
Le tableau ci-dessous résume l’utilisation du disque et les temps d’exécution présentés dans ce chapitre.
<!-- paragraph -->
```{r}
data.table(
kb.dt,
construct.seconds=c(
timing.incomplete.construct[["elapsed"]],
timing.for.construct[["elapsed"]],
timing.named.construct[["elapsed"]]),
compile.seconds=as.numeric(c(
elapsed.incomplete,
elapsed.for,
elapsed.named)))
```
<!-- paragraph -->
Il ressort clairement du tableau de contingence ci-dessus que les `clickSelects` et `showSelected` nommés sont plus efficaces à ces deux égards et devraient être privilégiés aux boucles `for`.
<!-- paragraph -->
Exercices :
<!-- paragraph -->
- Utilisez des `clickSelects` et des `showSelected` nommés pour créer une visualisation illustrant le surapprentissage et le sous-apprentissage, comme nous l'avons fait dans [cette visualisation du modèle linéaire et des plus proches voisins](https://tdhock.github.io/2023-12-04-degree-neighbors/).
<!-- comment -->
- Utilisez des `clickSelects` et des `showSelected` nommés pour créer une visualisation de données dans votre domaine d’expertise.
<!-- paragraph -->
Dans le [chapitre 15](/ch15), nous vous expliquerons comment visualiser les algorithmes de recherche de racines (« zero-finding »).
<!-- paragraph -->